Datahåndering III: Statistikk#
Læringsutbytte
Etter å ha arbeidet med dette temaet, skal du kunne:
bruke deskriptiv statistikk til å beskrive data med tall og visualiseringer
utføre regresjonsanalyse og tolke resultatene
If your experiment needs a statistician, you need a better experiment.
—Fysikeren Ernest Rutherford (1871–1937)
Deskriptiv statistikk#
Vi kan bruke statistikk til å oppsummere, beskrive og tolke data. Vi skiller mellom to typer statistikk:
Deskriptiv statistikk: oppsummere og beskrive data
Statistisk interferens: trekke slutninger fra data
Vi skal mest se på deskriptiv statistikk her. La oss vende tilbake til pingvindataene våre. Vi kan få en enkel statistisk oversikt over datasettet vårt ved å bruke funksjonen describe.
import pandas as pd
pingvindata = pd.read_csv("data/penguins.txt", delimiter = ",")
pingvindata.describe()
| bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | |
|---|---|---|---|---|
| count | 342.000000 | 342.000000 | 342.000000 | 342.000000 |
| mean | 43.921930 | 17.151170 | 200.915205 | 4201.754386 |
| std | 5.459584 | 1.974793 | 14.061714 | 801.954536 |
| min | 32.100000 | 13.100000 | 172.000000 | 2700.000000 |
| 25% | 39.225000 | 15.600000 | 190.000000 | 3550.000000 |
| 50% | 44.450000 | 17.300000 | 197.000000 | 4050.000000 |
| 75% | 48.500000 | 18.700000 | 213.000000 | 4750.000000 |
| max | 59.600000 | 21.500000 | 231.000000 | 6300.000000 |
Vi kan forklare disse størrelsene slik:
count = antall registrerte tilfeller
mean = gjennomsnittet for alle individer
std = standardavviket (dette skal vi se nærmere på seinere)
min = minste registrerte verdi
25 % = “nedre kvartil”. Her har 25 % av pingvinene for eksempel en kroppsmasse mellom 3550 og minste verdi (2700).
50 % = “midtre kvartil”. Her har 50 % av pingvinene for eksempel en kroppsmasse mellom 3550 og 4050.
75 % = “øvre kvartil”. Her har 75 % av pingvinene for eksempel en kroppsmasse på 4750 eller under. Det betyr også at 25 % har en kroppsmasse på 4750 til maksverdien 6300.
max = høyeste registrerte verdi
Vi kan også finne mange av disse størrelsene ved å bruke ulike funksjoner:
Underveisoppgave
Prøv ut programmet ovenfor og utforsk de ulike variablene slik at du forstår hva alle de statistiske størrelsene betyr. Forklar begrepet kvartil.
Mål på spredning#
Det finnes mange måter å beskrive spredningen i en variabel på. Vi skal se på følgende måter:
Variasjonsbredde
Interkvartilbredde
Varians og standardavvik
Variasjonsbredde er et enkelt mål på spredning, nemlig differansen mellom største og minste verdi (\(x_{maks} - x_{min})\). De to andre målene må vi se litt nøyere på.
Underveisoppgave
Regn ut variasjonsbredden i nebbdybde hos pingvinene.
Interkvartilbredde#
Vi har sett hvordan vi kan regne ut kvartiler.
Kvartiler og persentiler
Kvartiler er fjerdedeler av et utvalg. For eksempel er nedre kvartil (\(Q_1\)) den høyeste verdien av en variabel som tilhører de 25 % minste verdiene innenfor en variabel. Midtre kvartil (\(Q_2\)) er verdien midt i datasettet, det vil si at 50 % av dataene ligger over og under dette punktet. Den midtre kvartilen kalles også for medianen. Tredje kvartil (\(Q_3\)) markerer verdien som 75 % av dataene ligger under (og de resterende 25 % av dataene ligger over).
Dersom vi bruker en vilkårlig prosent som ikke representerer en fjerdedel, kaller vi det en persentil.
Differansen mellom første og tredje kvartil er et godt mål på spredning. Dette er et mål på spredningen til de 50 % midtre dataene, og påvirkes derfor ikke av data som ligger langt unna de andre dataene (såkalte “uteliggere”). Dette målet kalles interkvartilbredde (IQR) eller bare kvartilbredde:
\(IQR = Q_3 - Q_1\)
Kvartiler kan visualiseres enkelt med et boksplott:
import seaborn as sns
sns.boxplot(data = pingvindata, x = "species", y = "bill_depth_mm")
<AxesSubplot:xlabel='species', ylabel='bill_depth_mm'>
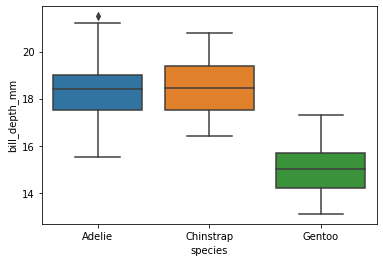
Vi kan forklare dette plottet slik:
Den svarte linja midt i boksene er medianen.
50 % av verdiene ligger innenfor boksene. Boksene markerer altså interkvartilområdet, fra \(Q_1\) til \(Q_3\).
Vi har to utstikkere på hver side av boksen. De viser \(IQR\cdot1.5 = 1.5(Q_3 - Q_1)\) i begge retninger.
Verdier som ligger utenfor disse utstikkerne, kalles uteliggere. Dette er unormalt store eller små verdier. Dette kan si oss noe om vi har mange unormale verdier eller om vi har noen få som vi kanskje kan fjerne ved nærmere statistisk analyse. Vi ser kun én uteligger hos Adelie-pingvinene, med unormalt dypt nebb.
Underveisoppgave
Lag et boksplott over kroppsmasse som funksjon av øya pingvinene er funnet på. Identifiser eventuelle uteliggere.
Underveisoppgave
sns.boxplot(data = pingvindata, x = "island", y = "body_mass_g")
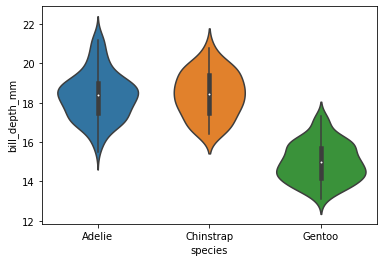
Et alternativ til boksplott er fiolinplott. Prinsippet er ganske likt som med boksplott, men det viser i tillegg fordelingen av de ulike verdiene ved å angi “tynne” og “tykke” områder. Medianen markeres med en hvit prikk, og IQR med en tykk, svart linje. Uteliggere markeres ofte ikke i fiolinplott.
sns.violinplot(data=pingvindata, x='species', y='bill_depth_mm')
<AxesSubplot:xlabel='species', ylabel='bill_depth_mm'>
Varians og standardavvik#
I motsetning til IQR, som tar utgangspunkt i medianen, tar varians og standardavvik utgangspunkt i gjennomsnittet. Varians (\(\sigma^2\)) er et mål på variasjonen i et datasett og skrives ofte som \(\sigma^2\). Det er definert som den gjennomsnittlige kvadrerte avstanden til gjennomsnittet av variabelen:
\(\sigma^2 = \frac{1}{n-1}\sum_{i=1}^n(x_i-\bar{x})^2\)
Her er n totalt antall målinger, \(x_i\) er hver måleverdi og \(\bar{x}\) er gjennomsnittet av alle måleverdier. Egentlig bør vi jo dele på \(n\) når vi skal finne gjennomsnittet av denne differansen. Men vi deler på \(n - 1\) istedenfor \(n\) fordi datasettet vårt inneholder et utvalg måledata fra en tenkt fullstendig populasjon. Når vi deler på \(n - 1\) får vi en større varians enn når vi deler på \(n\). En forklaring på dette er at dersom vi hadde samlet inn data fra hele populasjonen, eller gjort tusenvis av målinger av den samme parameteren, ville spredningen (variansen) av målingene vært litt mindre sammenliknet med når vi har færre datapunkter. Vi kaller derfor variansen som er definert ovenfor, for empirisk varians.
Kort oppsummert gjør vi følgende for å regne ut varians:
Finner differansen mellom hver måleverdi/hvert datapunkt og trekker fra gjennomsnittet. Dette gir avstanden mellom et målepukt og den gjennomsnittlige verdien.
Kvadrerer differansen for å kun få positive verdier. Om vi ikke hadde gjort dette, ville mange negative og positive verdier utlignet hverandre, slik at for eksempel to ekstreme verdier på hver sin side av gjennomsnittet ville gitt 0 i varians.
Summerer alle de kvadrerte differansene og deler på antallet målinger (her antallet målinger minus 1). Dette gir den gjennomsnittlige variansen for hele variabelen.
Siden vi kvadrerer avstanden mellom verdier og gjennomsnittet når vi regner ut varians, blir ofte variansen svært stor dersom det finnes noen verdier som ligger et stykke unna gjennomsnittet. I tillegg får den kvadrerte enheter (som \(g^2\) og \(mm^2\), og det er vanskelig å sammenlikne den med måledataene. Derfor er det vanligere å bruke standardavvik som et mål på spredning. Det er definert som den posive kvadratrota av variansen:
\(\sigma = \sqrt{\frac{1}{n-1}\sum_{i=1}^n(x_i-\bar{x})^2}\)
Vi kan enkelt finne variansen og standardavviket med Pandas. Pandas deler på \(n-1\), og ikke \(n\), slik som vi også har gjort.
varians = pingvindata["bill_depth_mm"].var()
standardavvik = pingvindata["bill_depth_mm"].std()
Underveisoppgave
Forklar hva varians og standardavvik er. Lag to Python-funksjoner som regner ut henholdsvis gjennomsnitt og standardavvik. Funksjonene skal gi samme svar som std- og mean-funksjonen i Pandas.
Visualisering av feil#
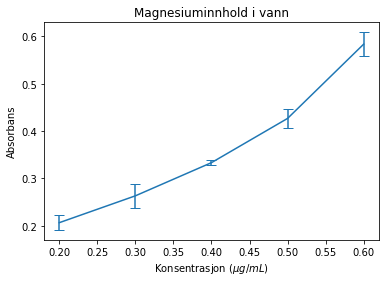
En enkel visualisering av standardavviket når vi gjør flere ulike målinger, er ved hjelp av usikkerhetsstolper. Usikkerhetsstolper kan vi bruke når vi har gjort flere målinger på samme variabel. Vi kan ta som eksempel et eksperiment der vi skal konstruere en standardkurve for magnesiumkonsentrasjonen i en vannprøve. Vi bruker en serie på 0.2, 0.3, 0.4, 0.5 og 0.6 \(\mu\)g/mL Mg\(^{2+}\) som vi analyserer tre ganger hver med et flammeatomabsorpsjonsspektrofotometer (for øvrig et nydelig ord!). Da har vi tre målinger for absorpsjon per konsentrasjon. Nedenfor ser du en måte å gjøre dette på. Vi bruker funksjonen errorbar som tar \(x\)-verdier, gjennomsnittet og standardavviket som parametre. I tillegg kan vi sette streker på toppen av feilen med capsize i en ønsket størrelse. Hvis vi ikke ønsker strekene mellom datapunktene i plottet ovenfor, kan vi legge inn argumentet fmt=’none’.
import numpy as np
import matplotlib.pyplot as plt
konsentrasjon = [0.2, 0.3, 0.4, 0.5, 0.6]
absorbans = [[0.21, 0.22, 0.19], [0.26, 0.29, 0.24], [0.33, 0.33, 0.34], [0.41, 0.42, 0.45], [0.56, 0.61, 0.58]]
standardavvik = []
snitt = []
for element in absorbans:
snitt.append(np.mean(element))
standardavvik.append(np.std(element, ddof = 1)) # ddof = 1 betyr at vi deler på n - 1.
plt.errorbar(konsentrasjon, snitt, yerr = standardavvik, capsize=5)
plt.errorbar
plt.xlabel('Konsentrasjon ($\mu g/mL$)')
plt.ylabel('Absorbans')
plt.title('Magnesiuminnhold i vann')
plt.show()
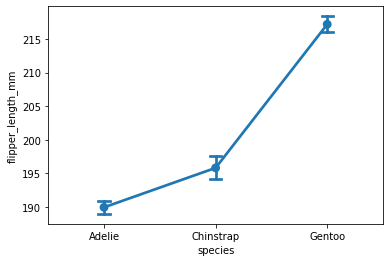
Det finnes også tilsvarende plott i seaborn-biblioteket, som er enklere å bruke dersom vi har dataene som en data frame:
sns.pointplot(data=pingvindata,x="species", y="flipper_length_mm", capsize=.1)
<AxesSubplot:xlabel='species', ylabel='flipper_length_mm'>
Regresjon#
Alle måledata er diskrete verdier. Det betyr at de er ikke-kontinuerlige, altså at det ikke finnes uendelig mange verdier mellom punktene vi har. Vi representerer diskrete data som punkter dersom vi plotter det, mens kontinuerlige data/funksjoner plotter vi som linjer og kurver. Noen ganger kan vi derimot lage kontinuerlige modeller som representerer de diskrete dataene våre. En slik modell kan beskrive både verdier imellom datapunktene, men også utenfor datasettet vi har samlet. En enkel form for modellering er regresjon.
Regresjon
Regresjon er en prosess der vi tilnærmer diskrete data med en kontinuerlig funksjonsmodell.
La oss ta et eksempel der vi har brukt et spektrometer til å måle absorbansen til ulike standardløsninger med permanganationer \(MnO_4^{-}\). Vi har gjort dette fordi vi har en løsning med ukjent innhold av permanganationer. Da trenger vi å gjøre en lineær regresjon for å kunne avgjøre hvilken konsentrasjon absorbansen til den ukjente prøva tilsvarer. Programmet nedenfor lager en standardkurve/kalibreringskurve med funksjonen polyfit fra numpy-biblioteket. Denne funksjonen bruker minste kvadraters metode til å tilpasse funksjonsverdier til datapunktene på en måte som gir minst mulig varians.
import numpy as np
import matplotlib.pyplot as plt
permanganat = [4.0, 8.0, 12.0, 16.0, 20.0] # konsentrasjon i ppm
absorbans = [0.18, 0.32, 0.51, 0.68, 0.85]
reg = np.polyfit(permanganat, absorbans, 1) # får koeffisientene a og b i en lineær regresjon (grad 1)
x = np.linspace(0, 25, 1000) # nye x-verdier for regresjonslinja
y = np.polyval(reg, x) # gir et uttrykk på formen y = ax + b, der x = permanganatkonsentrasjonen
plt.scatter(permanganat,absorbans,label='Datapunkter')
plt.plot(x, y, color = 'red',label='Tilpasset kurve')
plt.legend()
plt.title('Standardkurve')
plt.xlabel('Konsentrasjon av MnO$_4^-$ (ppm)')
plt.ylabel('Absorbans')
plt.grid()
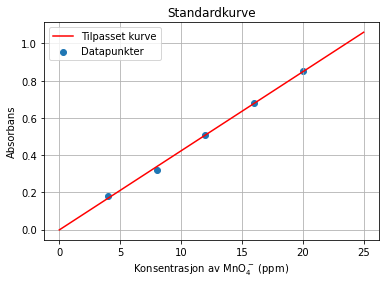
Funksjonen polyfit utfører polynomregresjon på dataene våre, og tar som argument \(x\)- og \(y\)-verdiene i datasettet, og deretter graden av polynomet. Her har vi brukt grad 1 for lineær regresjon. Dette gir en array med koeffisientene a og b for uttrykket \(y = ax + b\). Tilsvarende vil en andregradsregresjon gi koeffisientene a, b og c for \(ax^2 + bx + c\) og så videre. Vi kan dermed bruke disse koeffisientene videre til å lage nye \(y\)-verdier basert på de opprinnelige \(x\)-verdiene. Til det kan vi bruke funksjonen polyval, som tar to parametre: koeffisientene som er gitt av polyfit-funksjonen og de nye x-verdiene som vi skal regne ut funksjonsverdier til.
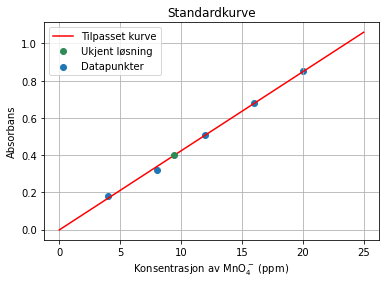
La oss si at den ukjente prøva med permanganationer ga en absorbans på 0.40. Vi kan bruke regresjonskurven vår til å finne hva slags konsentrasjon dette tilsvarer på følgende måte:
import numpy as np
import matplotlib.pyplot as plt
permanganat = [4.0, 8.0, 12.0, 16.0, 20.0] # konsentrasjon i ppm
absorbans = [0.18, 0.32, 0.51, 0.68, 0.85]
ukjent_abs = 0.40
# Gjør regresjonen
reg = np.polyfit(permanganat, absorbans, 1) # får koeffisientene a og b i en lineær regresjon (grad 1)
x = np.linspace(0, 25, 1000) # nye x-verdier for regresjonslinja
y = np.polyval(reg, x) # gir et uttrykk på formen y = ax + b, der x = permanganatkonsentrasjonen
# Regner ut den ukjente konsentrasjonen
ukjent_kons = (ukjent_abs - reg[1])/reg[0]
print("Konsentrasjonen til permanganat =", round(ukjent_kons,1), "ppm.")
plt.scatter(permanganat,absorbans,label='Datapunkter')
plt.plot(x, y, color = 'red',label='Tilpasset kurve')
plt.plot(ukjent_kons, ukjent_abs, linestyle = " ", marker = "o", color = 'seagreen', label = "Ukjent løsning")
plt.legend()
plt.title('Standardkurve')
plt.xlabel('Konsentrasjon av MnO$_4^-$ (ppm)')
plt.ylabel('Absorbans')
plt.grid()
Konsentrasjonen til permanganat = 9.5 ppm.
Underveisoppgave
Fritt klor (\(Cl_2\)) finnes i en del drikkevann fordi klorforbindelser blir tilsatt klorforbindelser for å desinfisere vannet. I programmet nedenfor har vi lagt inn en serie målinger som er gjort på standardløsninger av konsentrasjonen av fritt klor i vann gitt i ppm. Gjør en regresjon av dataene og lag en standardkurve. Regn ut konsentrasjonen av fritt klor i en drikkevannsprøve med absorbans 0.656 ppm.
Løsningsforslag
import numpy as np
import matplotlib.pyplot as plt
klor = [0, 0.50, 1.00, 1.50, 2.00] # konsentrasjon i ppm
absorbans = [0.000, 0.270, 0.543, 0.813, 1.084]
ukjent_abs = 0.656
# Gjør regresjonen
reg = np.polyfit(klor, absorbans, 1) # får koeffisientene a og b i en lineær regresjon (grad 1)
x = np.linspace(0, 2.2, 1000) # nye x-verdier for regresjonslinja
y = np.polyval(reg, x) # gir et uttrykk på formen y = ax + b, der x = permanganatkonsentrasjonen
# Regner ut den ukjente konsentrasjonen
ukjent_kons = (ukjent_abs - reg[1])/reg[0]
print("Konsentrasjonen til fritt klor =", round(ukjent_kons,2), "ppm.")
plt.scatter(klor,absorbans,label='Datapunkter')
plt.plot(x, y, color = 'red',label='Tilpasset kurve')
plt.plot(ukjent_kons, ukjent_abs, linestyle = " ", marker = "o", color = 'seagreen', label = "Ukjent løsning")
plt.legend()
plt.title('Standardkurve')
plt.xlabel('Konsentrasjon av Cl$_2$ (ppm)')
plt.ylabel('Absorbans')
plt.grid()
plt.show()
Når vi har utført regresjon, kan vi også forutsi datapunkter som er utenfor datapunktene våre. Dette kaller vi ekstrapolering. Ved å ekstrapolere kan vi forutsi hvordan et system har vært eller kommer til å bli. En skal derimot være svært forsiktig med å trekke slutninger basert på ekstrapolering! Det kan likevel være en god indikasjon på trender og utviklingen i et system.
Vi kan også lage visualiseringer med automatiske regresjonskurver. La oss vende tilbake til pingvinene våre. For å se om det kan være noen sammenhenger som er verdt å lage en modell av, kan vi enkelt visualisere mange ulike variabler mot hverandre ved å bruke et parplott (pairplot) fra seaborn-biblioteket:
import seaborn as sns
sns.pairplot(data = pingvindata, hue = "species")
<seaborn.axisgrid.PairGrid at 0x28083e73220>
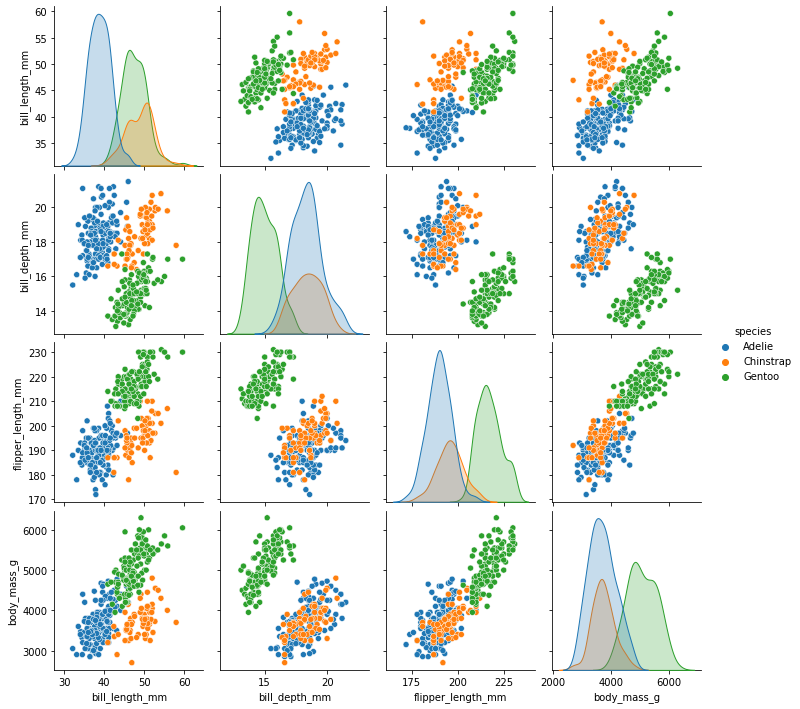
Vi tar utgangspunkt i kroppsmasse og vingelengde, som ser ut til å være lineært avhengig av hverandre. Et plott med regresjonslinje kan vi lage ved å bruke funksjonen lmplot (eller regplot) fra seaborn-biblioteket:
sns.lmplot(data = pingvindata, x = "body_mass_g", y = "flipper_length_mm")
<seaborn.axisgrid.FacetGrid at 0x280879f8430>
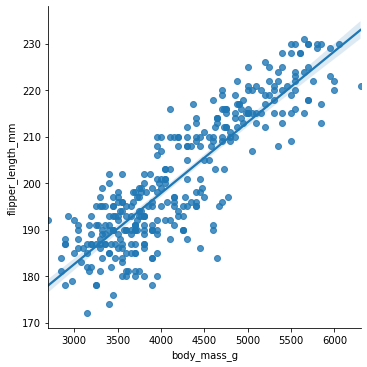
Vi ser at vi får en regresjonslinje med et svakt blått område på hver side av linja. Dette området indikerer usikkerheten i verdiene. Desto større dette området er, desto mer usikkert er det at regresjonslinja er en god modell i dette området. Vi kan gjøre dette tydeligere ved å velge to variabler som ikke varierer så godt lineært:
sns.lmplot(data = pingvindata, x = "bill_depth_mm", y = "flipper_length_mm")
<seaborn.axisgrid.FacetGrid at 0x28087a06e20>
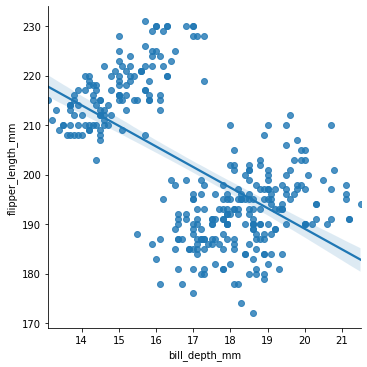
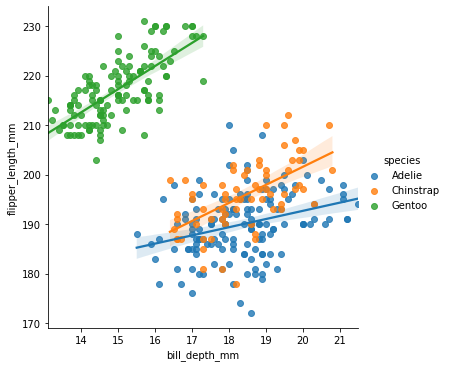
Vi kan også gjøre regresjon på ulike variabler, slik som ulike arter, for seg:
sns.lmplot(data = pingvindata, x = "bill_depth_mm", y = "flipper_length_mm", hue = "species")
<seaborn.axisgrid.FacetGrid at 0x280864bb970>
Korrelasjon#
Korrelasjoner kan brukes til å analysere sammenhenger mellom parametre. Korrelasjon beregnes som en verdi mellom -1 og 1. En verdi som brukes ofte som mål på korrelasjon, er Pearsons korrelasjonskoeffisient. Positiv korrelasjon betyr at det er en proporsjonal sammenheng mellom parametrene, og en negativ korrelasjon betyr at det er en omvendt proporsjonal sammenheng. Desto større absoluttverdien til korrelasjonen er, desto større sammenheng er det mellom parametrene. Vi kan visualisere korrelasjon gjennom et korrelasjonsplott:
corr = pingvindata.corr() # Regner ut korrelasjonskoeffisientene
sns.heatmap(corr, annot=True) # Lager visualisering. Parameteren annot = True gir tallene i boksene.
plt.xticks(rotation=45) # Roterer x-akseteksten 45 grader
plt.show()
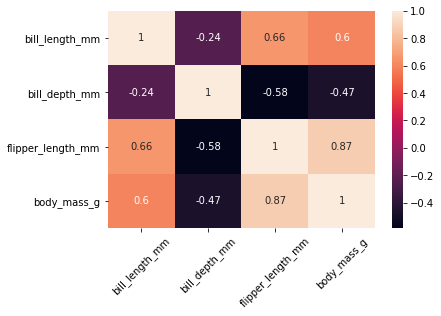
pingvindata.corr()
| bill_length_mm | bill_depth_mm | flipper_length_mm | body_mass_g | |
|---|---|---|---|---|
| bill_length_mm | 1.000000 | -0.235053 | 0.656181 | 0.595110 |
| bill_depth_mm | -0.235053 | 1.000000 | -0.583851 | -0.471916 |
| flipper_length_mm | 0.656181 | -0.583851 | 1.000000 | 0.871202 |
| body_mass_g | 0.595110 | -0.471916 | 0.871202 | 1.000000 |
Vi ser for eksempel at det er ganske stor positiv korrelasjon mellom nebblengde og lengden på vingene (luffene). Det betyr at dersom en pingvin har langt nebb, har den ofte også lange vinger. Det er derimot negativ korrelasjon mellom nebbdybde og vingelengde, som betyr at desto dypere nebb, desto kortere vinger har pingvinen.
Det er derimot viktig å ikke blande korrelasjon med årsak-virkning (kausalitet). Den ene tingen forklarer ikke nødvendigvis den andre (selv om det kan gjøre det), det er bare en sammenheng mellom parametrene.
Underveisoppgave
I programmet nedenfor finner du datasettet “iris.txt”. Les inn datasettet og gjør en korrelasjonsanalyse av variablene. Hvilke faktorerer korrelerer positivt og negativt med hverandre? Finnes det logiske forklaringer på det?
Løsningsforslag
import pandas as pd
import matplotlib.pyplot as plt
import seaborn as sns
iris = pd.read_csv("iris.txt")
corr = iris.corr()
sns.heatmap(corr, annot=True,)
plt.xticks(rotation=45)
plt.show()
Videoer#
I videoen nedenfor kan du få en innføring eller repetisjon i den grunnleggende teorien bak regresjonsanalyse:
